library(raster)
dem<- raster ("Capas/dem/DEM.tif")
plot(dem)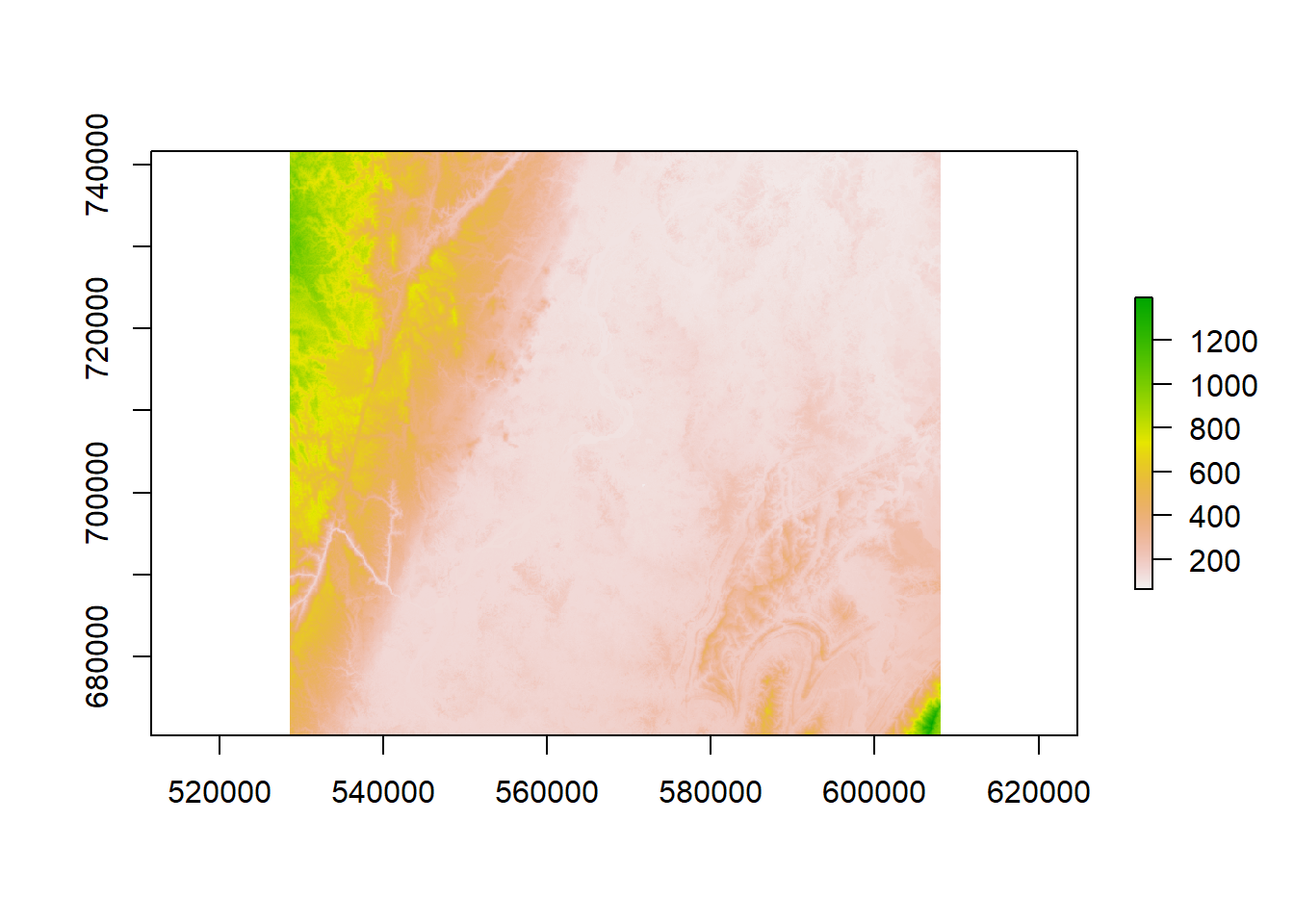
Daniela O
October 1, 2024
class : RasterLayer
dimensions : 5705, 6368, 36329440 (nrow, ncol, ncell)
resolution : 12.5, 12.5 (x, y)
extent : 528403.3, 608003.3, 670363, 741675.5 (xmin, xmax, ymin, ymax)
crs : +proj=utm +zone=18 +datum=WGS84 +units=m +no_defs
source : DEM.tif
names : DEM DEM
Min. 86
1st Qu. 130
Median 163
3rd Qu. 293
Max. 1405
NA's 0Los stack nos permiten incluir varios raster en solo uno. Ten en cuenta que los raster tengan la misma proyección y el mismo extent
# Cargar cada raster de forma independiente
bio1<-raster("Capas/bio1.tif")
bio2<-raster("Capas/bio2.tif")
bio3<-raster("Capas/bio3.tif")
bio4<-raster("Capas/bio4.tif")
# Crear un stack
bios<-stack(bio1, bio2, bio3, bio4)
plot(bios)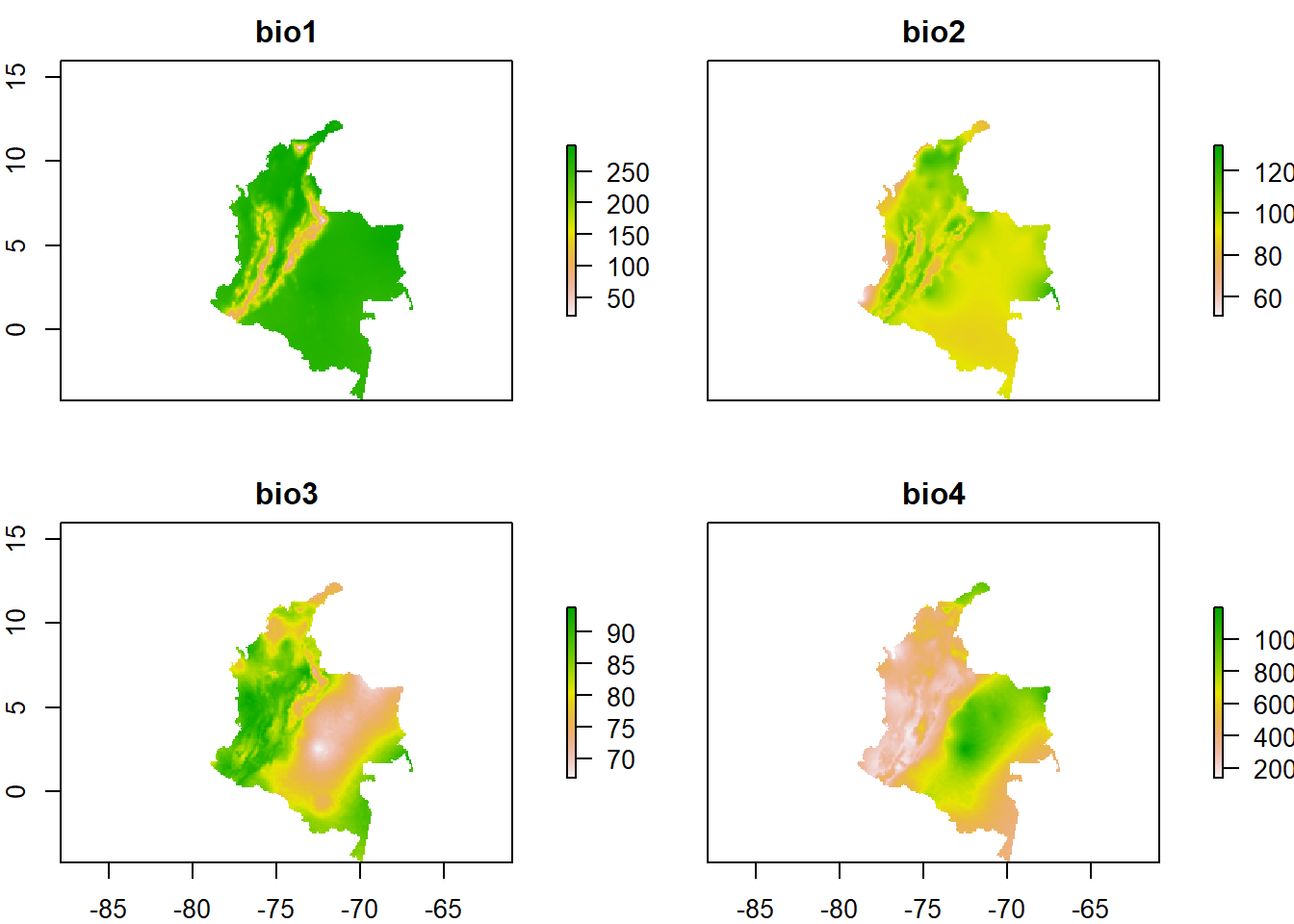
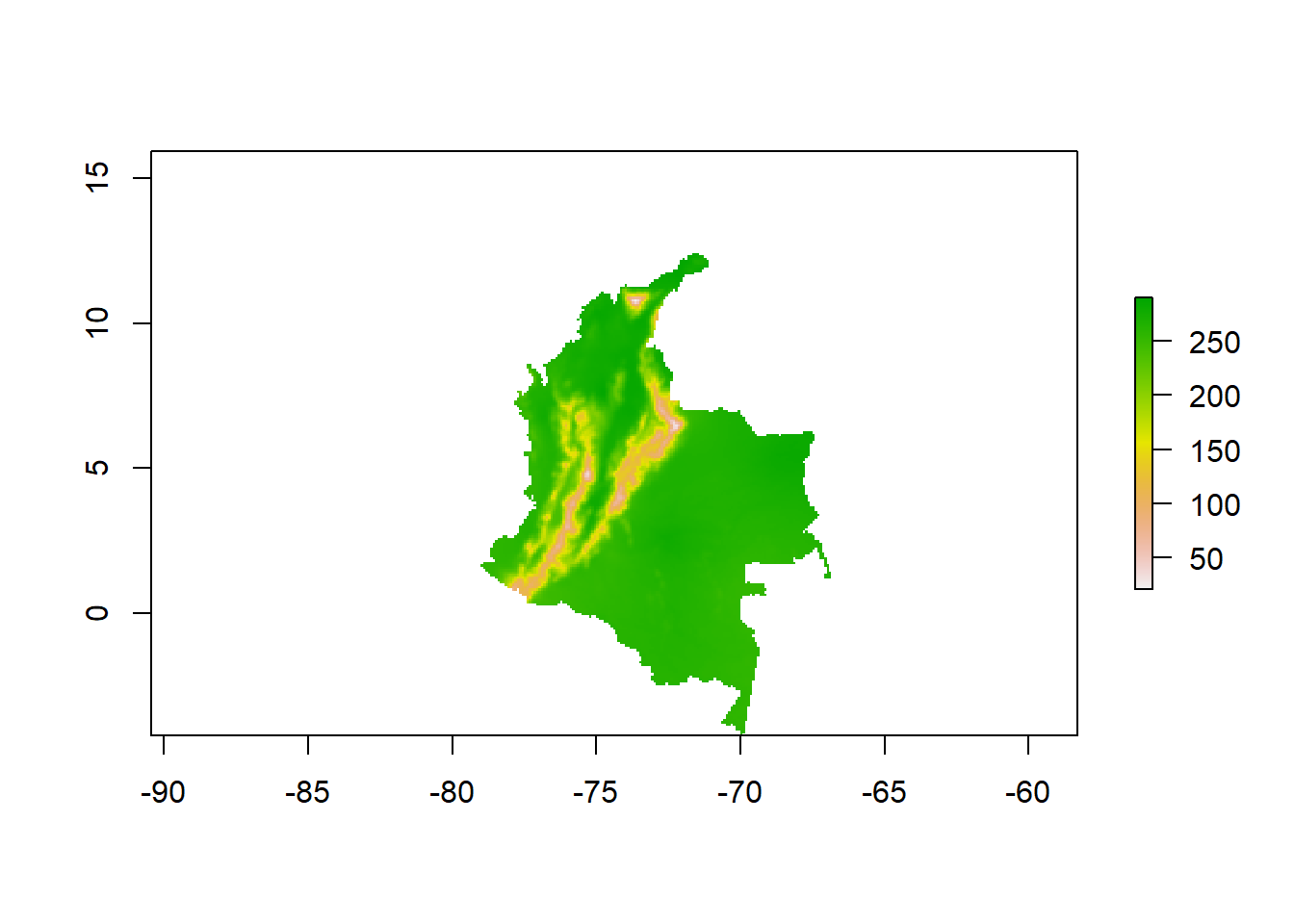
# Cargar todos los raster de una carpeta en un stack
bios19<-stack(list.files(path="Capas/", pattern = ".tif", full.names = TRUE))
bios19class : RasterStack
dimensions : 242, 180, 43560, 19 (nrow, ncol, ncell, nlayers)
resolution : 0.0833, 0.0833 (x, y)
extent : -81.8726, -66.8786, -4.2123, 15.9463 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +no_defs
names : bio1, bio10, bio11, bio12, bio13, bio14, bio15, bio16, bio17, bio18, bio19, bio2, bio3, bio4, bio5, ...
min values : 21.154696, 28.154696, 9.340766, 268.150604, 86.975685, 0.000000, 11.293017, 171.408234, 0.000000, 23.518999, 0.000000, 51.086884, 67.000000, 140.313873, 74.712906, ...
max values : 290.19424, 301.67709, 282.98999, 9912.64258, 1049.16125, 653.17615, 115.61718, 3034.19897, 2161.18262, 2428.07837, 2785.71558, 131.88080, 93.79576, 1193.03503, 362.45981, ... 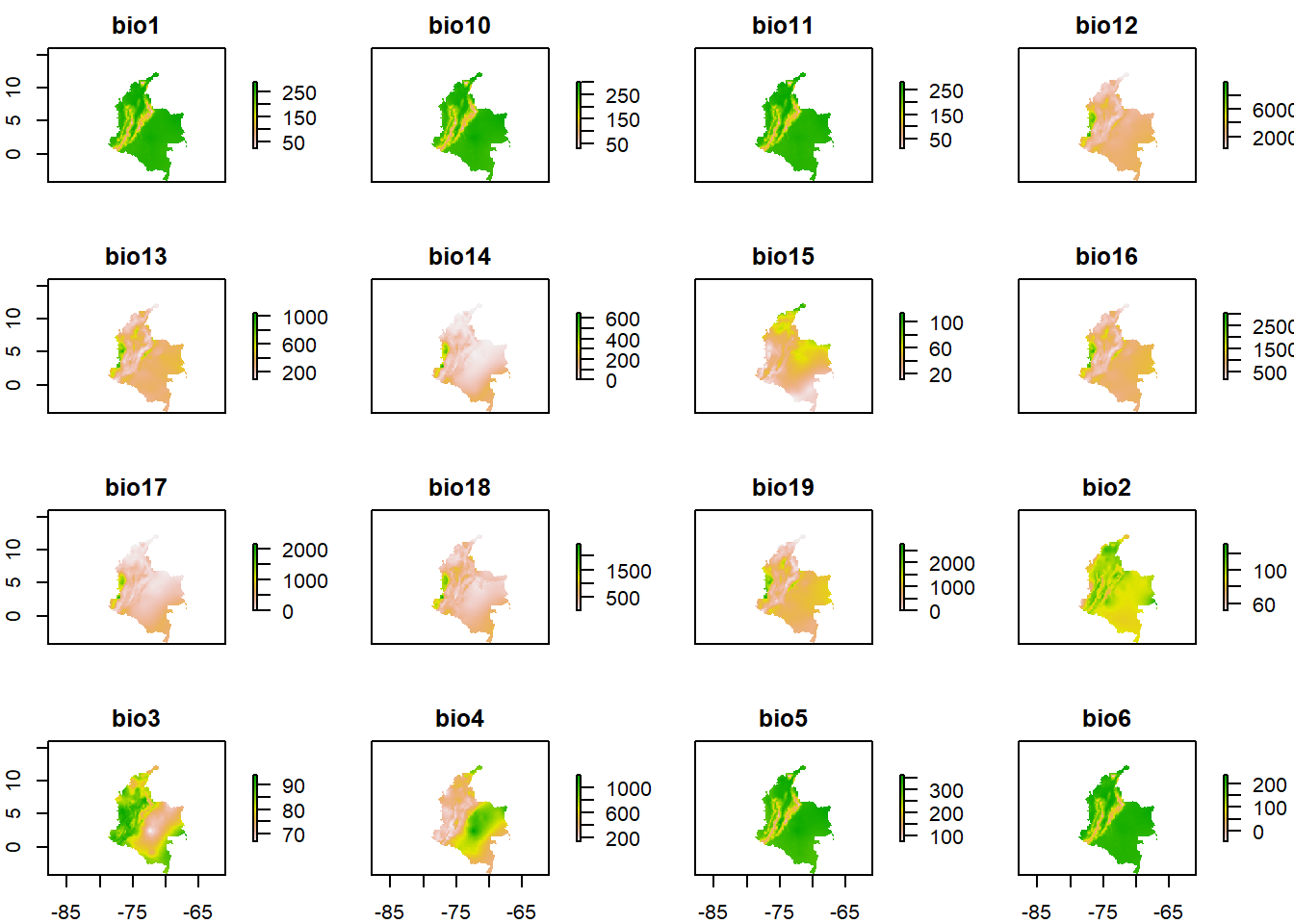
Se puede hacer para un raster o un stack
b1<- raster("Capas/bio1.tif")
# reproyectar un solo raster
b1utm<-projectRaster(b1, crs="+proj=utm +zone=18 +ellps=WGS84 +datum=WGS84 +units=m +no_defs")
b1utmclass : RasterLayer
dimensions : 254, 191, 48514 (nrow, ncol, ncell)
resolution : 9220, 9210 (x, y)
extent : -312700.6, 1448319, -513239.8, 1826100 (xmin, xmax, ymin, ymax)
crs : +proj=utm +zone=18 +datum=WGS84 +units=m +no_defs
source : memory
names : bio1
values : 30.55129, 290.1524 (min, max)# Creo stack
b12<-stack(b1, b2)
# Rreproyectar un stack
b12_utm<-projectRaster(b12, crs="+proj=utm +zone=18 +ellps=WGS84 +datum=WGS84 +units=m +no_defs")
b12_utmclass : RasterBrick
dimensions : 254, 191, 48514, 2 (nrow, ncol, ncell, nlayers)
resolution : 9220, 9210 (x, y)
extent : -312700.6, 1448319, -513239.8, 1826100 (xmin, xmax, ymin, ymax)
crs : +proj=utm +zone=18 +datum=WGS84 +units=m +no_defs
source : memory
names : bio1, bio2
min values : 30.55129, 51.27524
max values : 290.1524, 131.3687